Les mouvements inédits de l’ADN dans des milieux nanométriques
Confiné dans un espace réduit à quelques nanomètres lorsqu’il se réplique, l’ADN bouge alors d’une manière extrêmement difficile à observer. Une collaboration franco-nippone a montré pour la première fois que l’ADN nanoconfiné suivait un mouvement balistique, au lieu du mouvement brownien classique qui anime habituellement les particules. Publiés dans ACS Nano, ces travaux impliquent que l’ADN confiné dans les engins biomoléculaires comme lors de sa réplication, dépense moins d’énergie que prévu.
Le mouvement brownien décrit les trajectoires aléatoires prises par une particule sous l’effet de chocs avec des particules plus petites, telles que les molécules d’eau ou de gaz qui constituent son environnement. En l’absence de collision, on parle de mouvement balistique, qui ne peut donc se vérifier que dans l’infime intervalle de temps entre deux chocs. Des chercheurs du Laboratory for integrated micro mechatronics systems (LIMMS, CNRS/Université de Tokyo), du Laboratoire d’électrochimie moléculaire (LEM, CNRS/Université Paris Cité) et de la NTT Corporation (Japon) ont pour la première fois montré l’existence d’un mouvement brownien balistique de l’ADN retenu dans un espace nanométrique. Cette configuration, où la chaine d’ADN est enfermée dans une cavité de 1,5 à 5 nanomètres, correspond à la manière dont il est naturellement confiné lors du processus de réplication.
La littérature considérait jusqu’alors que l’ADN nanoconfiné était trop peu raide et trop peu affecté par l’inertie pour présenter un mouvement balistique. Pour étudier plus en détail la question, l’équipe a développé le logiciel Q-biol, qui décrit la dynamique moléculaire d’une chaine d’ADN piégée entre deux électrodes planes très rapprochées et les transferts d’électrons, et confronté ces simulations à des données expérimentales. Le brin d’ADN se voit adjoindre un petit marqueur moléculaire ferrocène, transporteur d’électrons et suffisamment léger pour ne pas perturber ses déplacements. En fonction des mouvements du brin d’ADN, le ferrocène entre en collision avec les électrodes formant la cavité, et y transfère un électron, si bien que le courant véhiculé à travers la nanocavité est proportionnel à la fréquence des mouvements de l’ADN et à sa raideur. Suivre les électrons, qui se déplacent à très grande vitesse, permet notamment de caractériser le phénomène à l’échelle de la nanoseconde. Les prédictions de Q-biol, confirmées par l’expérience, indiquent qu’une fois confiné dans un espace inférieur à 5 nanomètres, l’ADN entre dans un régime balistique.
Publiés dans ACS Nano, ces travaux apportent une meilleure compréhension de la consommation énergétique des machines biomoléculaires impliquant l’ADN. L’adoption d’un régime balistique suppose en effet que l’ADN se meut en transférant moins d’énergie à l’environnement, puisqu’il percute moins de particules, que ce qui était jusqu’alors considéré. Sachant que la réplication de l’ADN représente 13 % de la consommation de glucose de certaines cellules, il se pourrait que les dimensions des nanocavités naturelles dans lesquelles ce régime est possible aient été optimisées au fil de l’évolution pour dépenser moins d’énergie. L’équipe compte aussi rendre librement accessible le programme Q-biol afin qu’il serve aux chercheurs qui utilisent l’ADN dans des biocapteurs, notamment pour la détection de cellules cancéreuses.
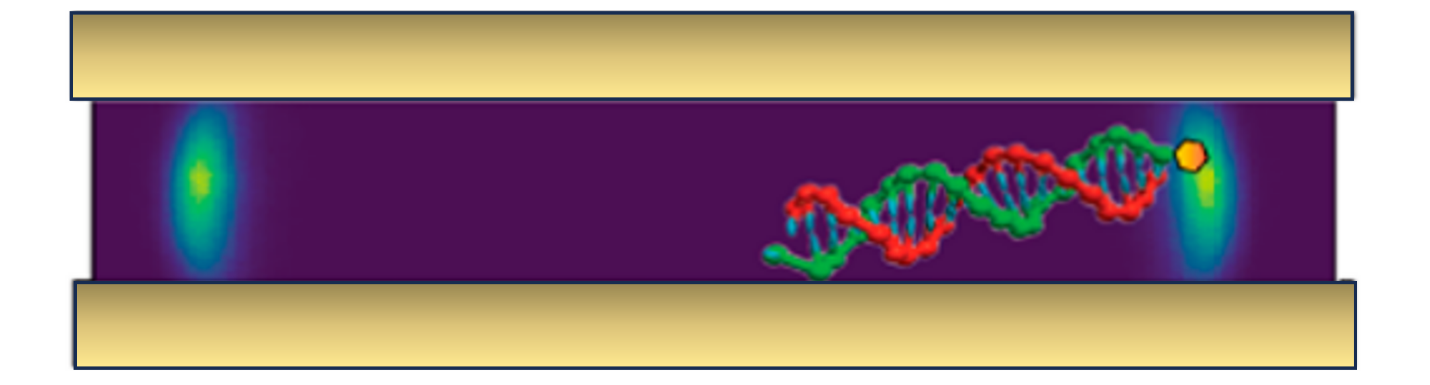
© Nicolas Clément et Christophe Demaille
Références
Ballistic Brownian Motion of Nanoconfined DNA
Ignacio Madrid, Zhiyong Zheng, Cedric Gerbelot, Akira Fujiwara, Shuo Li, Simon Grall, Katsuhiko Nishiguchi, Soo Hyeon Kim, Arnaud Chovin, Christophe Demaille, and Nicolas Clement.
ACS Nano, 2023
https://doi.org/10.1021/acsnano.3c04349